新闻网讯(通讯员基医)2021年9月,武汉大学基础医学院梁凯威教授课题组在基因组学期刊Genome Research发表题为“Ligand-induced native G-quadruplex stabilization impairs transcription initiation”的论文,并被选为当期的封面。该研究结合G-四链体抗体BG4和CUT&Tag技术,建立了一种灵敏的天然G-四链体全基因组检测方法G4-CUT&Tag;进一步结合转录组学和蛋白组学分析,发现了G-四链体配体诱导的G-四链体结构稳定能够快速地改变染色质状态,阻碍通用转录因子与启动子区域的结合,阐述了G-四链体抑制基因转录起始的新机制。
据了解,基因组的大部分DNA以经典的双链B型DNA形式存在,但是也存在一系列的非经典DNA二级结构,例如G-四链体(G4)和R-Loop(R-环)。G-四链体是富含鸟嘌呤的单链核酸折叠形成的一种非经典核酸二级结构,它们在进化上保守,广泛分布于端粒、基因启动子、转录因子结合位点等区域。R-Loop是一种特殊的三链核酸结构,是转录过程中新生的RNA链侵入DNA双链导致形成DNA-RNA杂交双链和一条游离的DNA单链。非经典DNA二级结构约占基因组的3-5%,它们被认为参与了基因转录、DNA复制等多种基本生命过程,并与神经退行性疾病、肿瘤等人类重大疾病密切相关。
此外,今年2月,梁凯威课题组在Science Advances发表题为“Genomic profiling of native R loops with a DNA-RNA hybrid recognition sensor”的研究论文,该研究构建了一个新的DNA-RNA杂交体识别因子GST-His6-2xHBD,并结合CUT&Tag技术开发了一种R-Loop原位定位新方法:R-Loop CUT&Tag,并且进一步系统比较了GST-His6-2xHBD和R-Loop识别抗体S9.6,在体外富集和原位捕获定位策略下,获得了R-Loop在基因组上的分布,并且发现不同R-Loop定位方法产生的R-Loop图谱的差异主要由不同的R-Loop捕获策略所导致,而非识别因子本身,解释了R-Loop基因组研究中长期存在的争议。
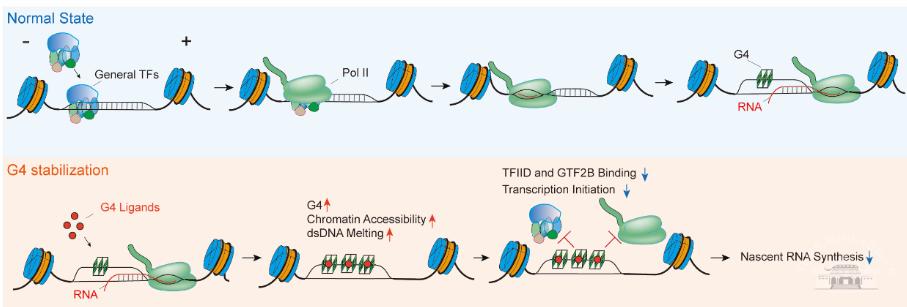
综上,上述研究提供了灵敏的天然非经典DNA原位检测方法,并揭示了非经典DNA结构、染色质状态、基因转录之间的调控机制,不仅为非经典核酸二级结构的研究提供了新思路,还深化了对G-四链体和R-Loop生物学功能及其作为DNA靶向治疗靶点的应用潜力的理解。
武汉大学基础医学院博士研究生李聪慧、王红红、尹智囊和房萍萍副研究员为G-四链体相关研究论文的共同第一作者,武汉大学基础医学院梁凯威教授为该文通讯作者。武汉大学基础医学院硕士研究生王康和博士研究生王红红、李聪慧为R-Loop相关研究论文的共同第一作者,梁凯威教授和房萍萍副研究员为该文共同通讯作者。武汉大学生命科学学院的黄健教授、陈亮研究员以及化学与分子学院的翁小成教授对研究均有重要贡献。两项研究均获得了科技部重点专项青年项目、基金委青年项目、国家青年人才引进项目及武汉大学人才引进项目的资助。
论文链接:
https://www.genome.org/cgi/doi/10.1101/gr.275431.121
https://advances.sciencemag.org/content/7/8/eabe3516